Bacterias promotoras
Bacterias promotoras de crecimiento vegetal (BPCV)
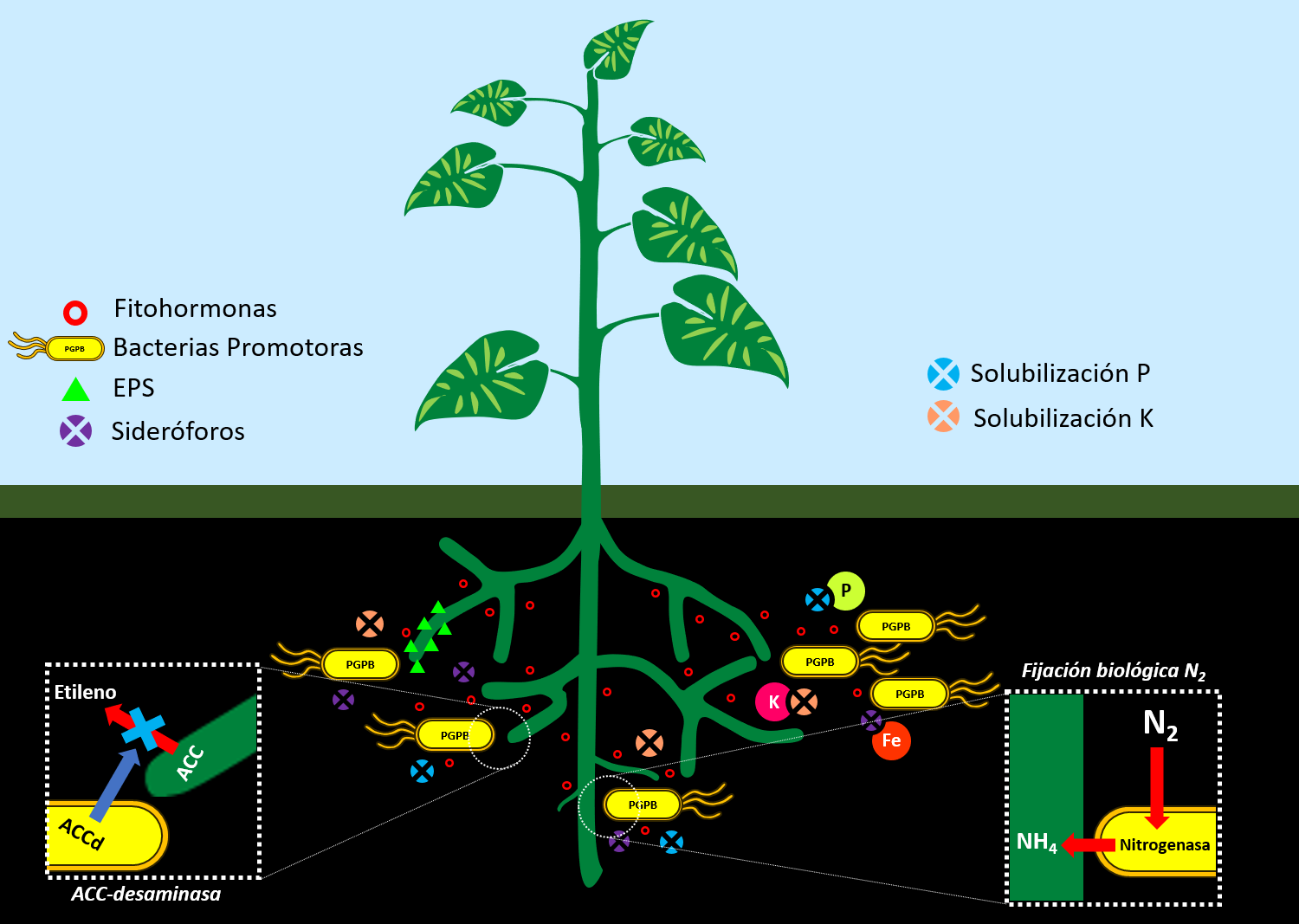
Las interacciones planta-microorganismo-suelo-ambiente (Figura 1) son esenciales para el desarrollo de las especies vegetales y su productividad. Las denominadas bacterias promotoras de crecimiento vegetal (BPCV) tienen la capacidad de ayudar a su huésped a mejorar la disponibilidad de nutrientes en el suelo, regulando su crecimiento y/o protegiéndolo en presencia de diversos tipos de estreses (Souza et al., 2015).
Este grupo de microorganismos posee varios mecanismos, algunos de ellos enunciados en este catálogo, entre los cuales se destacan: la fijación biológica de nitrógeno, la mineralización de fósforo orgánico e inorgánico, la producción de fitohormonas, de exopolisacáridos, la inducción de resistencia sistémica y la mitigación de estreses bióticos y abióticos.
Muchas de estas actividades funcionan de manera simultánea: las bacterias pueden mejorar la adquisición de nutrientes en las plantas y al mismo tiempo, pueden protegerlas del ataque de patógenos y de condiciones ambientales adversas (Vimal et al., 2017).
1. Biofertilización
La biofertilización se entiende como la aplicación de microorganismos que promueven el crecimiento de las plantas al incrementar el acceso al suministro de nutrientes (Gaiero et al., 2013). Esta práctica es utilizada actualmente como complemento a la fertilización química en diferentes cultivos, con el objetivo de reducir la aplicación de productos de síntesis.
1.1. Fijación biológica de Nitrógeno
La fijación biológica de nitrógeno se refiere a la conversión de nitrógeno atmosférico a amonio por parte de un grupo específico de bacterias que cuentan con la presencia de la enzima nitrogenasa y forman nódulos (Figura 2) (Gaiero et al., 2013). Este grupo de miroorganismos es uno de los más utilizados como principios activos en biofertilizantes comerciales. Lo componen algunos géneros como Rhizobium, Azospirillum, Herbaspirillum, Azotobacter, Azoarcus, entre otros.

La relación de las bacterias con su huésped se puede clasificar de acuerdo a su proximidad en tres categorías: asociación libre (bacterias de vida libre), asociacion endofítica (bacterias que se encuentran en espacios intercelulares, normalmente en la rizósfera), y endosimbiosis (bacterias que se encuentran en membranas derivadas de las plantas) (Paramasivan et al., 2016). La endosimbiosis rizobio-leguminosa es de vital importancia en cultivos como soya, frijol y arveja debido a que gran parte del nitrógeno requerido por la planta es obtenida a través de esta simbiosis aportando hasta un 80% de este elemento. Actualmente existen en el mercado productos con base en bacterias de los géneros Rhizobium, Bradyrhizobium y Mesorhizobium, (VIMAL et al., 2017).
1.1.1 Actividad de Reducción de Acetileno
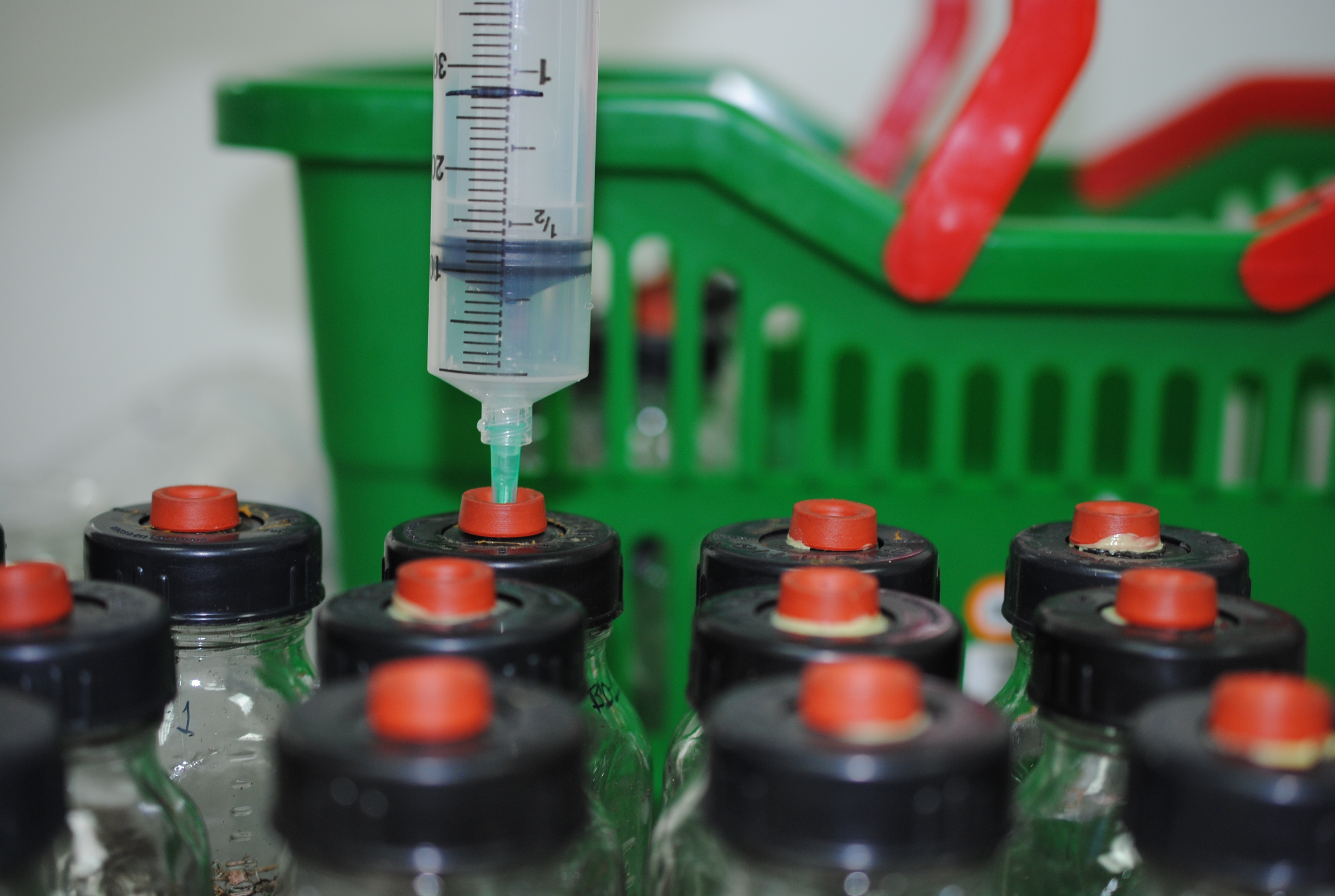
Las raíces noduladas se introducen en frascos herméticamente cerrados con un volumen de 280 mL a los cuales se le sustituye el 10% (v/v) de la atmósfera (28 mL) con acetileno y se incuban durante una hora a 32° C (Figura 3). La concentración de etileno se cuantifica inyectando 1 mL de la atmósfera del frasco del cultivo en un cromatógrafo de gases (Agilent Technologies GC-FID 7890a); columna HayeSep N (20 pies, 1/8 pulgada, 2 mm) flujo N2 20 mLmin-1 (gas arrastre). El diseño estadístico que se utiliza es completamente al azar en donde cada tratamiento (accesión) tiene como mínimo 3 repeticiones, además se establece un control negativo, es decir sin inoculación (Grove & Malajczuk, 1987).
1.2 Solubilización de Fósforo
La solubilización de fósforo es la actividad metabólica que transforma el fósforo (P) insoluble tanto inorgánico como orgánico en formas asimilables para las plantas. Las bacterias llevan a cabo la solubilización de P en los suelos mediante los siguientes mecanismos: acidificación del medio, formación de complejos metálicos, reducción de metales y disolución de fosfatos por medio de enzimas (Pii et al., 2015).
El impacto de la solubilización de P en un país como Colombia, que tiene deficiencias hasta del 98% de este macronutriente, es significativo. Dentro de sus principales efectos se encuentran: 1. la disminución de la dosis de fertilizantes minerales fosfatados en cultivos como algodón, arroz, tomate, maíz, soja, trigo, cacahuate, nuez, entre otros (Alori et al., 2017; Mehta et al., 2015; Bakhshandeh et al., 2017; Anzuay et al., 2017, Bonilla y Morales, 2005), 2. mitigación de los efectos del estrés por sequía en las plantas (Behera et al., 2014) y 3. su gran influencia en la fitorremediación de metales pesados en los suelos (Ahemad, 2015).
1.2.1 Determinación de solubilización de Fósforo
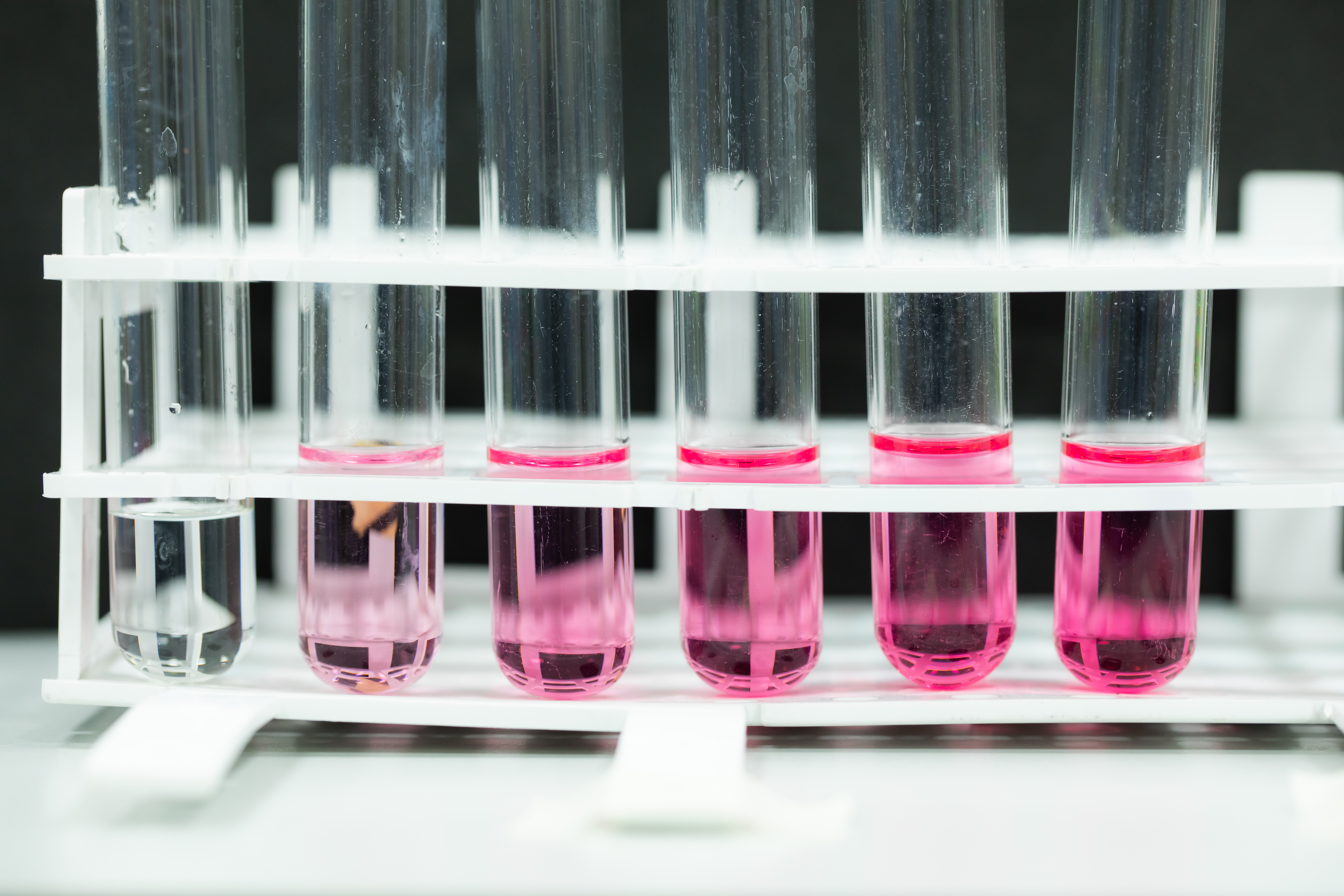
La determinación del P liberado por las cepas se lleva a cabo en el medio de cultivo SRS líquido (Composición g L-1: KH2PO4 0.2, K2HPO4 0.8, MgSO4.7H2O 0.2, CaSO4 0.1, MoO3 0.001, Sacarosa 5.0, FeSO4.7H2O 0.04) (Sundara y Sinha, 1963) suplementado con fosfato tricalcico (Ca3 (PO4)2, fosfato de hierro (FePO4) o fosfato de aluminio (AlPO4) 5% w/v). En todos los casos los ensayos se realizan por triplicado. Los inóculos se ajustan en una solución de NaCl (0,85% m/v) DO600 = 0,500 (aproximadamente 108 células mL-1). Luego se inocula 1mL de la suspensión bacteriana en 20mL de los medios SRS en Erlenmeyer de 125mL. La incubación se lleva a cabo a 30 ± 2 °C por 12 días con agitación constante a 180 xg (Estrada et al., 2013). Al final de este periodo el P soluble se cuantifica empleando el método de azul de fosfo-molibdeno usando una absorbancia de 712 nm (Murphy y Riley, 1962; Watanabe y Olsen, 1965). Previamente se realiza una curva estándar utilizando concentraciones conocidas de 20, 40, 60, 80 y 100 mg L-1 de K2HPO4. (Figura 4).

1.3. Solubilización de Potasio (K)
El Potasio (K), es uno de los tres elementos esenciales para el crecimiento y buen desarrollo de las plantas; se encuentra disponible como: i) iones de potasio (K+) en la solución del suelo, ii) firmemente retenido en las superficies de los minerales arcillosos y la materia orgánica, iii) fuertemente retenidos o iV) fijados por minerales micáceos degradados. La forma más común de potasio en el suelo es el feldespato y mica con 90 a 98% (McAfee, 2008). Solamente el uno o dos por ciento de este elemento se encuentra disponible para las plantas (Etesami et al., 2018).
El potasio desempeña un papel indispensable en la síntesis proteica, fotosíntesis, activación enzimática por parte de la planta, regula la permeabilidad de la membrana celular y la apertura estomática; de igual forma le confiere a la planta resistencia a enfermedades y ataque de insectos (Rehm y Schmitt, 2002; Almeida et al., 2015; Cecílio Filho et al., 2015; Gallegos-Cedillo et al., 2016; Hussain et al., 2016; White and Karley, 2010; Yang et al., 2018, Verma and Palani, 1997). Existe gran cantidad de microrganismos en el suelo que son capaces de solubilizar formas “no disponibles” de potasio mediante la liberación de ácidos orgánicos (Groudev, 1987; Ullman et al., 1996), dentro de los géneros de hongos y bacterias con capacidad solubilizadora de potasio se han reportado, Pseudomonas, Bacillus, Rhizobium y Aspergillus (Prajapati et al., 2013).
1.3.1 Solubilización cualitativa de potasio (K)

Las cepas son crecidas en medio Aleksandrov (Composición en g.L-1: 5.0 de C6H12O6, 0.5 de MgSO4, 0.005 de FeCl3, 0.1 de CaCO3, 2 de Ca₃(PO₄)₂ modificado (Hu et al., 2006), mediante siembra por agotamiento. La modificación consiste en usar oxalato de calcio –CaOx- (4g.L-1) como única fuente de carbono. Además, se evalúan tres fuentes insolubles de potasio: i. biotita, ii. moskovita y iii. feldespato en polvo (0.09% w/v para cada caso) (Figura 5).
2. Producción de reguladores del crecimiento vegetal
Los reguladores de crecimiento vegetal o “fitohormonas” son compuestos naturales o sintéticos que afectan procesos metabólicos o de desarrollo en las plantas superiores (Rademacher, 2015). Se encuentran clasificados principalmente en 5 grupos – auxinas, giberelinas, etileno, ácido abscísico y citoquinas-, pero en la actualidad se pueden incluir además los brasinosteroides y jasmonatos. Estas sustancias además incrementan la resistencia de las plantas a diversos factores ambientales ya que pueden inducir o suprimir la expresión de una amplia gama de genes (Spence and Bais, 2015).
Las auxinas son uno de los reguladores de crecimiento vegetal más estudiados, siendo el ácido indol acético (AIA) la principal auxina en plantas. Estos compuestos han sido asociados con procesos como gravitropismo y fototropismo, diferenciación del tejido vascular, dominancia apical, formación de raíces laterales y adventicias, estimulación de la división celular y elongación de tallo y raíces (Cassán et al., 2014). Algunas bacterias tienen la capacidad de sintetizar bajas cantidades de fitohormonas afectando no sólo el desarrollo de su huésped, sino la respuesta a estreses bióticos y abióticos. Algunos de los géneros más reportados son Pseudomonas, Bacillus, Azospirillum, Burkholderia, entre otros (Egamberdieva et al., 2017).
2.1 Producción de índoles totales
A partir de la fermentación líquida en medio de cultivo suplementado con triptófano (100 ug mL-1), se toma 1 mL de cultivo, se centrifuga a 16000 xg durante 6 min, se toman 100 microlitros del sobrenadante y se llevan a reacción con 100 microlitros del reactivo de Salkowsky modificado sobre placas ELISA. La reacción es incubada por 30 min en completa oscuridad por triplicado. Una vez pasado el tiempo de reacción, se determina la absorbancia en un espectrofotómetro a 540 nm. Con los datos de absorbancia se obtiene el promedio de las réplicas y se reemplazan los datos, en la ecuación de la recta de la curva patrón para determinar la producción de indoles totales (Carreño et al., 2000; Glickmann and Dessaux, 1995). Previamente se realiza una curva estándar utilizando concentraciones conocidas de 0, 10, 20, 30, 40, 50, 60, 70, 80, 90, 100, 120, 140, 160, 180 y 200 mg L-1 de Ácido 3 Indolacético (Figura 6).
3. Producción de sideróforos
El hierro es un elemento esencial para el crecimiento de las plantas. Debido a su rol como cofactor en muchos procesos metabólicos, su deficiencia puede llevar a la disrupción de procesos tan importantes como la respiración y la fotosíntesis (Radzki et al., 2013).
Bajo condiciones limitantes de hierro, algunas plantas y bacterias producen agentes quelantes o sideróforos, compuestos de bajo peso molecular que mediante la mineralización o el secuestro de este elemento, lo hacen disponible para el desarrollo y crecimiento de las plantas. Los sideróforos se pueden clasificar de acuerdo con sus grupos funcionales como hidroxamatos, catecolatos y carboxilatos. Algunas bacterias tienen la capacidad de producir sideróforos compuestos por varios grupos funcionales, como es el caso de las pioverdinas producidas por las bacterias del género Pseudomonas (Ahmed y Holmström, 2014).
3.1 Determinación de sideróforos
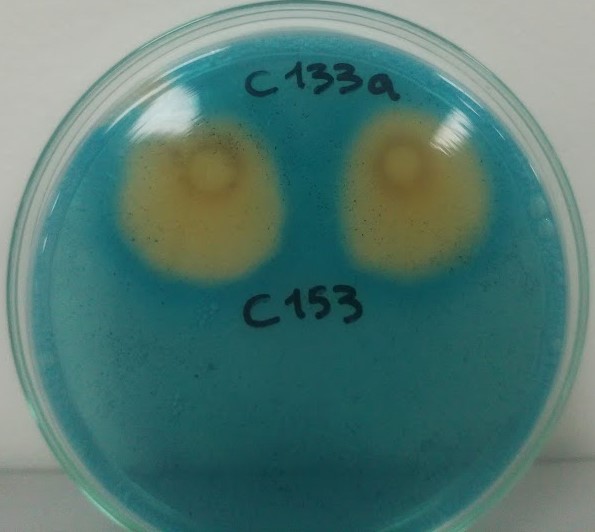
Las accesiones son sembradas en medio de cultivo CAS y posteriormente incubadas a 28 ± 20C verificando el cambio de color del medio de cultivo, dependiendo de la velocidad de crecimiento de cada accesión, por un tiempo de 15 días (Lauden et al., 2011) (Figura 7).
4. Actividad de la enzima ACC-desaminasa
Diversos tipos de estrés biótico (plagas y enfermedades) y abiótico (déficit nutricional, sequía, salinidad) desencadenan la producción de altos niveles de etileno en las plantas, afectando sensiblemente el desarrollo vegetal (Glick et al., 2014). Algunas bacterias promotoras de crecimiento vegetal poseen la capacidad de disminuir los niveles de etileno en las plantas mediante la actividad de la enzima 1-aminocilopropano-1-carboxilato (ACC) desaminasa (ACCd) (Glick, 2014). Esta enzima es responsable de la ruptura del precursor del etileno en plantas, el ACC, en amonio y α-cetobutirato (Honma y Shimomura, 1978).
El uso de PGPB con actividad ACC-desaminasa se ha documentado en cultivos como Ají disminuyendo el impacto de sequía y salinidad (Maxton et al., 2018). Microorganismos como Azospirillum sp., Bacillus sp., Rhizobium sp., y Pseudomonas sp., han demostrado ser efectivos en la mitigación del déficit hídrico en cultivos de maíz, trigo, soya, lavanda y plantas modelo como Arabidopsis (Vurukonda et al., 2016). Además, el uso de rizobios con esta actividad en leguminosas aumenta la eficiencia y velocidad de nodulación sumado a su mitigación de estreses abióticos (Nascimento et al., 2018).
4.1 Evaluación cualitativa de ACC-desaminasa
Los aislamientos se crecen en caldo nutritivo (NB), LB o TSB por 48h a 28 ± 2 ° C (el tiempo y la temperatura depende del microorganismo). Luego tomando una alícuota en un microtubo de 1,5mL centrifugar a 14000rpm x 5min y re suspender en MgSO 4 0.1M realizando 3 lavados volviendo a centrifugar con el objetivo de remover todo el medio de cultivo restante y con ello cualquier traza de nitrógeno posible. Después de los lavados, debe hacerse una dilución de 10-veces en MgSO 40.1M estéril, este será el inóculo de células lavadas para el proceso. Debe prepararse una solución de ACC 3mM estéril mediante filtración por membrana (0,2µm) y almacenarla a -20 ° C hasta su uso. La solución debe ser descongelada momento anterior a su uso, debido a que el ACC en solución acuosa a temperatura ambiente tiende a volatilizarse.
En una placa de 96 pozos estéril, se adicionan por pozo 120µL de medio mínimo de sales (MSM) o M9. A las primeras cuatro líneas adicionar 15µL de MgSO 4 0.1M, a las siguientes 4 líneas 15µL (NH4) 2 SO 4 0.1M y finalmente a las 4 líneas restante 15µL de ACC 3mM recién descongelado. La bacteria diluida 10-veces en MgSO 4 0.1M se adiciona en relación de 15µL en cada pozo (exceptuando el blanco). Las soluciones de las fuentes de nitrógeno pueden prepararse y esterilizarse mediante filtración por membrana (0,2µm) en recipientes estériles al momento de preparar la placa. No debe olvidarse incluir un control de ACC-deaminasa positivo como lo es la Pseudomonas putida UW4.
Las placas se llevan a incubar a 28 ± 2 ° C durante 48h (el tiempo y la temperatura depende del microorganismo) en agitación orbital en un espectrofotómetro de microplacas midiendo la OD = 600nm. Los valores DO obtenidos en los pozos de ACC- y (NH 4 ) 2 SO 4 - deben ser comparados con el de MgSO 4 para determinar la habilidad de los aislamientos PGPR de utilizar ACC para su crecimiento.
5. Síntesis de exopolisacáridos (EPS)
Los exopolisacáridos (EPS) abarcan una amplia gama de polímeros de carbohidratos con aspecto mucilaginoso que son secretados en el ambiente por enzimas bacterianas (Birch et al., 2019). Los EPS tienen diversas estructuras y propiedades físicas que son empleadas en diferentes industrias teniendo una amplia aplicación biotecnológica, como la industria farmacéutica, procesos industriales alimenticios, tratamientos de aguas residuales y de biorremediación (Shukla and Dave, 2018). Su importancia radica en los siguientes efectos: Mejoran la estructura del suelo mediante la agregación de este a través de microorganismos, protegen a las bacterias de diversos estreses ambientales (sequía, salinidad, temperatura y metales pesados), influyen en la mitigación de los efectos del estrés por sequía en las plantas, protegen las células de compuestos antimicrobianos y anticuerpos y permiten la adhesión a raíces, tejidos animales y otras bacterias (Subair et al., 2015).
5.1 Determinación cualitativa de exopolisacaridos (EPS).
Para el análisis de la determinación cualitativa de exopolisacáridos se producen 5 ml de biomasa, hasta la fase exponencial del microorganismo o durante 5 días, en medio líquido TSB bajo condiciones estándar. Se adiciona 0.2 ml de EDTA 0.26 M y 0.1 ml de NaCl 5 M al cultivo bacteriano en un falcón de 15 ml y se centrifuga la mezcla a 4800 xg durante 10 minutos. Posteriormente, se transfiere el volumen del sobrenadante a otro falcón y se adiciona etanol helado en una relación 2:1 respecto al sobrenadante. No mezclar. Se deja durante 24 h a 4ºC sin agitación.
La centrifugación se realiza a 4600 rpm durante 10 minutos y se elimina el sobrenadante. Se adiciona agua desionizada al EPS precipitado hasta completar volumen inicial (~ 5 ml) (Figura 8). Se homogeniza mediante el uso del vortex. Se toman 0.3 ml de la muestra y se adicionan 0.3 ml de fenol 5%. Se añaden 1.5 ml de ácido sulfúrico concentrado. Esta reacción se deja estabilizar durante 30 min en un baño maría a 30°C. Si se manejan volúmenes más pequeños, se debe tener en cuenta que la relación general de la reacción es 1:1:5 por muestra:fenol:ácido sulfúrico, respectivamente (Bradford, 1976; DuBois et al., 1956).

6. Caracterización genómica de las cepas
La calidad de los datos brutos se revisa utilizando FastQC (Andrews, 2010), las lecturas de baja calidad se eliminan utilizando 'ILLUMINACLIP', un programa de Trimmomatic v0.32 (Bolger et al., 2014). Las lecturas de alta calidad son ensambladas en contigs usando IDBA-UD (Peng et al., 2012), luego se realiza la predicción de genes usando Prodigal (Hyatt et al., 2010) e inmediatamente, usando MyTaxa (Luo et al., 2014) se hace el mapeo taxonómico. A partir de estos resultados se infieren las asociaciones evolutivas con genomas cercanos completos almacenados en NCBI (Sayers et al., 2018), para ello se sigue la metodología descrita por Eren (2020) empleando para el análisis la colección de genes de Campbell et al. (2011) dentro de un perfil hmm por defecto en la plataforma de visualización Anvi´o (Eren et al., 2015), adicionalmente, utilizando FastANI (Jain et al., 2018), se calcula el valor ANI (Average Nucleotide Identity) con el fin de complementar los resultados inferenciales. Normalmente, los valores del ANI entre los genomas de una misma especie son superiores al 95% y valores entre el 80% y el 95% indican especies estrechamente relacionadas (Konstantinidis and Tiedje, 2005). Los resultados son graficados usando el paquete gplots v3.0.3 y el software estadístico R, versión 3.6.1 (Warnes et al., 2016). Por otro lado, se construyen bases de datos de secuencias de proteínas asociadas a marcadores moleculares de funciones biofertilizantes de interés como la fijación biológica de nitrógeno (nifK, nifQ y nifH), actividad ACC-desaminasa (acdR, LRP, FNR, CRP y acdS), la solubilización de fósforo (agp, ansB, aphA, Appa-phy, gdh y los genes del operón pqq), producción de indoles (I3AN, I3PAP, IAP y TrypP) y la producción de giberelinas (GA20-ox, GA7-ox, KAO, KO y KS), también se usan todos los genes reportados para el ciclo del nitrógeno (Tu et al., 2018). Por cada grupo de secuencias, se realiza un alineamiento múltiple por medio de MUSCLE (Edgar, 2004), inmediatamente, se construye un perfil hmm por grupo usando Hmmer (Eddy, 1998) y una vez construido este perfil se ejecuta la concatenación de todos perfiles. Luego, con cada uno de los genomas se realiza una búsqueda de homología dentro de este perfil hmm para detectar los genes marcadores, la búsqueda se realiza con un filtro de e value de 0.001. Finalmente, una vez identificados los genes de interés por genoma, empleando la herramienta PathVisio (Kutmon et al., 2015) se dibujan las rutas específicas de las funciones de interés biofertilizante a partir de las rutas de señalización de KEGG (Ogata et al., 1998).
Bibliografía
Ahemad, M., 2015. Phosphate-solubilizing bacteria-assisted phytoremediation of metalliferous soils: a review. 3 Biotech 5, 111-121.
Ahmed, E., Holmström, S.J.M., 2014. Siderophores in environmental research: roles and applications. Microbial Biotechnology 7, 196-208.
Almeida, H.J., Pancelli, M.A., Prado, R.M., Cavalcante, V.S., Cruz, F.J.R., 2015. Effect of potassium on nutritional status and productivity of peanuts in succession with sugar cane. Journal of soil science and plant nutrition 15, 1-10.
Alori, E.T., Glick, B.R., Babalola, O.O., 2017. Microbial phosphorus solubilization and its potential for use in sustainable agriculture. Frontiers in Microbiology 8.
Alves, G.C., Videira, S.S., Urquiaga, S., Reis, V.M., 2015. Differential plant growth promotion and nitrogen fixation in two genotypes of maize by several Herbaspirillum inoculants. Plant and Soil 387, 307-321.
Andrews, S., 2010. FastQC: a quality control tool for high throughput sequence data. Babraham Bioinformatics, Babraham Institute, Cambridge, United Kingdom.
Anzuay, M.S., Ciancio, M.G.R., Ludueña, L.M., Angelini, J.G., Barros, G., Pastor, N., Taurian, T., 2017. Growth promotion of peanut (Arachis hypogaea L.) and maize (Zea mays L.) plants by single and mixed cultures of efficient phosphate solubilizing bacteria that are tolerant to abiotic stress and pesticides. Microbiological Research 199, 98-109.
Bakhshandeh, E., Pirdashti, H., Lendeh, K.S., 2017. Phosphate and potassium-solubilizing bacteria effect on the growth of rice. Ecological Engineering 103, 164-169.
Baldani, J.I., Rouws, L., Cruz, L.M., Olivares, F.L., Schmid, M., Hartmann, A., 2014a. The Family Oxalobacteraceae. In: Rosenberg, E., DeLong, E.F., Lory, S., Stackebrandt, E., Thompson, F., Eds.), The Prokaryotes: Alphaproteobacteria and Betaproteobacteria. Springer Berlin Heidelberg, Berlin, Heidelberg, pp. 919-974.
Baldani, J.I., Videira, S.S., dos Santos Teixeira, K.R., Reis, V.M., de Oliveira, A.L.M., Schwab, S., de Souza, E.M., Pedraza, R.O., Baldani, V.L.D., Hartmann, A., 2014b. The Family Rhodospirillaceae. In: Rosenberg, E., DeLong, E.F., Lory, S., Stackebrandt, E., Thompson, F., Eds.), The Prokaryotes: Alphaproteobacteria and Betaproteobacteria. Springer Berlin Heidelberg, Berlin, Heidelberg, pp. 533-618.
Bashan, Y., de-Bashan, L.E., 2010. How the plant growth-promoting bacterium Azospirillum promotes plant growth—a critical assessment. In: Sparks, D.L., (Ed.), Advances in Agronomy. Academic Press, pp. 77-136.
Behera, M.D., Patidar, N., Chitale, V.S., Behera, N., Gupta, D., Matin, S., Tare, V., Panda, S.N., Sen, D.J., 2014. Increase in agricultural patch contiguity over the past three decades in Ganga River Basin, India. Current Science 107, 502-511.
Birch, J., Van Calsteren, M.-R., Pérez, S., Svensson, B., 2019. The exopolysaccharide properties and structures database: EPS-DB. Application to bacterial exopolysaccharides. Carbohydrate Polymers 205, 565-570.
Bolger, A.M., Lohse, M., Usadel, B., 2014. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics 30, 2114-2120.
Bonilla, B., Morales, G., 2005. Monibac: un biofertilizante con base en cepas nativas de Azotobacter sp. para incrementar la productividad y sostenibilidad del algodonero. Innovación y cambio tecnológico 4, 30-34.
Bradford, M.M., 1976. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry 72, 248-254.
Campbell, B.J., Yu, L., Heidelberg, J.F., Kirchman, D.L., 2011. Activity of abundant and rare bacteria in a coastal ocean. Proceedings of the National Academy of Sciences 108, 12776.
Carareto Alves, L.M., de Souza, J.A.M., Varani, A.d.M., Lemos, E.G.d.M., 2014. The Family Rhizobiaceae. In: Rosenberg, E., DeLong, E.F., Lory, S., Stackebrandt, E., Thompson, F., Eds.), The Prokaryotes: Alphaproteobacteria and Betaproteobacteria. Springer Berlin Heidelberg, Berlin, Heidelberg, pp. 419-437.
Cassán, F., Vanderleyden, J., Spaepen, S., 2014. Physiological and agronomical aspects of phytohormone production by model plant-growth-promoting rhizobacteria (PGPR) belonging to the genus Azospirillum. Journal of Plant Growth Regulation 33, 440-459.
Cecílio Filho, A.B., Feltrim, A.L., Mendoza Cortez, J.W., Gonsalves, M.V., Pavani, L.C., Barbosa, J.C., 2015. Nitrogen and potassium application by fertigation at different watermelon planting densities. Journal of soil science and plant nutrition 15, 928-937.
Chang, W.-T., Chen, Y.-C., Jao, C.-L., 2007. Antifungal activity and enhancement of plant growth by Bacillus cereus grown on shellfish chitin wastes. Bioresource Technology 98, 1224-1230.
DuBois, M., Gilles, K.A., Hamilton, J.K., Rebers, P.A., Smith, F., 1956. Colorimetric Method for Determination of Sugars and Related Substances. Analytical Chemistry 28, 350-356.
Eddy, S.R., 1998. Profile hidden Markov models. Bioinformatics 14, 755-763.
Edgar, R.C., 2004. MUSCLE: multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Research 32, 1792-1797.
Egamberdieva, D., Wirth, S.J., Alqarawi, A.A., Abd_Allah, E.F., Hashem, A., 2017. Phytohormones and beneficial microbes: Essential components for plants to balance stress and fitness. Frontiers in microbiology 8, 2104.
Eren, A.M., 2020. An anvi'o workflow for phylogenomics. In: LAB, M., (Ed.).
Eren, A.M., Esen, Ö.C., Quince, C., Vineis, J.H., Morrison, H.G., Sogin, M.L., Delmont, T.O., 2015. Anvi’o: an advanced analysis and visualization platform for ‘omics data. PeerJ 3, e1319.
Estrada, G.A., Baldani, V.L.D., de Oliveira, D.M., Urquiaga, S., Baldani, J.I., 2013. Selection of phosphate-solubilizing diazotrophic Herbaspirillum and Burkholderia strains and their effect on rice crop yield and nutrient uptake. Plant and Soil 369, 115-129.
Etesami, H., 2018. Bacterial mediated alleviation of heavy metal stress and decreased accumulation of metals in plant tissues: Mechanisms and future prospects. Ecotoxicology and Environmental Safety 147, 175-191.
Fibach-Paldi, S., Burdman, S., Okon, Y., 2012. Key physiological properties contributing to rhizosphere adaptation and plant growth promotion abilities of Azospirillum brasilense. FEMS Microbiology Letters 326, 99-108.
Gaiero, J.R., McCall, C.A., Thompson, K.A., Day, N.J., Best, A.S., Dunfield, K.E., 2013. Inside the root microbiome: Bacterial root endophytes and plant growth promotion. American Journal of Botany 100, 1738-1750.
Gallegos-Cedillo, V.M., Urrestarazu, M., Álvaro, J.E., 2016. Influence of salinity on transport of Nitrates and Potassium by means of the xylem sap content between roots and shoots in young tomato plants. Journal of soil science and plant nutrition 16, 991-998.
Glick, B.R., 2014. Bacteria with ACC deaminase can promote plant growth and help to feed the world. Microbiological Research 169, 30-39.
Glickmann, E., Dessaux, Y., 1995. A critical examination of the specificity of the salkowski reagent for indolic compounds produced by phytopathogenic bacteria. Applied and Environmental Microbiology 61, 793-796.
Groudev, S.N., 1987. Use of heterotrophic microorganisms in mineral biotechnology. Acta Biotechnologica 7, 299-306.
Grove, T.S., Malajczuk, N., 1987. Nitrogen fixation (acetylene reduction) by forest legumes: sensitivity to pre‐harvest and assay conditions. New Phytologist 106, 115-127.
Hu, X., Chen, J., Guo, J., 2006. Two Phosphate- and Potassium-solubilizing Bacteria Isolated from Tianmu Mountain, Zhejiang, China. World Journal of Microbiology and Biotechnology 22, 983-990.
Hungria, M., Campo, R.J., Souza, E.M., Pedrosa, F.O., 2010. Inoculation with selected strains of Azospirillum brasilense and A. lipoferum improves yields of maize and wheat in Brazil. Plant and Soil 331, 413-425.
Hussain, S., Khan, F., Cao, W., Wu, L., Geng, M., 2016. Seed Priming alters the production and detoxification of reactive oxygen intermediates in rice seedlings grown under sub-optimal temperature and nutrient supply. Frontiers in plant science 7, 439.
Hyatt, D., Chen, G.-L., LoCascio, P.F., Land, M.L., Larimer, F.W., Hauser, L.J., 2010. Prodigal: prokaryotic gene recognition and translation initiation site identification. BMC Bioinformatics 11, 119.
Idriss, E.E., Makarewicz, O., Farouk, A., Rosner, K., Greiner, R., Bochow, H., Richter, T., Borriss, R., 2002. Extracellular phytase activity of Bacillus amyloliquefaciens FZB45 contributes to its plant-growth-promoting effect Microbiology 148, 2097-2109.
Jain, C., Rodriguez-R, L.M., Phillippy, A.M., Konstantinidis, K.T., Aluru, S., 2018. High throughput ANI analysis of 90K prokaryotic genomes reveals clear species boundaries. Nature communications 9, 5114-5114.
Konstantinidis, K.T., Tiedje, J.M., 2005. Genomic insights that advance the species definition for prokaryotes. Proceedings of the National Academy of Sciences of the United States of America 102, 2567.
Kumar, P., Dubey, R.C., Maheshwari, D.K., 2012. Bacillus strains isolated from rhizosphere showed plant growth promoting and antagonistic activity against phytopathogens. Microbiological Research 167, 493-499.
Kutmon, M., van Iersel, M.P., Bohler, A., Kelder, T., Nunes, N., Pico, A.R., Evelo, C.T., 2015. PathVisio 3: An Extendable Pathway Analysis Toolbox. PLOS Computational Biology 11, e1004085.
Laranjo, M., Alexandre, A., Oliveira, S., 2014. Legume growth-promoting rhizobia: An overview on the Mesorhizobium genus. Microbiological Research 169, 2-17.
Logan, N.A., Vos, P.D., 2015. Bacillaceae. Bergey's Manual of Systematics of Archaea and Bacteria. John Wiley & Sons, Inc, pp. 1-1.
Luo, C., Rodriguez-R, L.M., Konstantinidis, K.T., 2014. MyTaxa: an advanced taxonomic classifier for genomic and metagenomic sequences. Nucleic Acids Research 42, e73-e73.
Maxton, A., Singh, P., Masih, S.A., 2018. ACC deaminase-producing bacteria mediated drought and salt tolerance in Capsicum annuum. Journal of Plant Nutrition 41, 574-583.
McBride, M.J., 2014. The Family Flavobacteriaceae. In: Rosenberg, E., DeLong, E.F., Lory, S., Stackebrandt, E., Thompson, F., Eds.), The Prokaryotes: Other Major Lineages of Bacteria and The Archaea. Springer Berlin Heidelberg, Berlin, Heidelberg, pp. 643-676.
Mehta, P., Walia, A., Kulshrestha, S., Chauhan, A., Shirkot, C.K., 2015. Efficiency of plant growth-promoting P-solubilizing Bacillus circulans CB7 for enhancement of tomato growth under net house conditions. Journal of Basic Microbiology 55, 33-44.
Mu’minah, Baharuddin, Subair, H., Fahruddin, 2015. Isolation and screening bacterial exopolysaccharide (EPS) from potato rhizosphere in highland and the potential as a producer indole acetic acid (IAA). Procedia Food Science 3, 74-81.
Murphy, J., Riley, J.P., 1962. A modified single solution method for the determination of phosphate in natural waters. Analytica Chimica Acta 27, 31-36.
Mus, F., Crook, M.B., Garcia, K., Garcia Costas, A., Geddes, B.A., Kouri, E.D., Paramasivan, P., Ryu, M.-H., Oldroyd, G.E.D., Poole, P.S., Udvardi, M.K., Voigt, C.A., Ané, J.-M., Peters, J.W., 2016. Symbiotic Nitrogen Fixation and the Challenges to Its Extension to Nonlegumes. Applied and Environmental Microbiology 82, 3698.
Nascimento, F.X., Tavares, M.J., Rossi, M.J., Glick, B.R., 2018. The modulation of leguminous plant ethylene levels by symbiotic rhizobia played a role in the evolution of the nodulation process. Heliyon 4, e01068.
Niu, D.-D., Liu, H.-X., Jiang, C.-H., Wang, Y.-P., Wang, Q.-Y., Jin, H.-L., Guo, J.-H., 2011. The plant growth–promoting rhizobacterium Bacillus cereus AR156 induces systemic resistance in arabidopsis thaliana by simultaneously activating Salicylate- and Jasmonate/Ethylene-dependent signaling pathways. Molecular Plant-Microbe Interactions® 24, 533-542.
Ogata, H., Goto, S., Fujibuchi, W., Kanehisa, M., 1998. Computation with the KEGG pathway database. Biosystems 47, 119-128.
Peng, Y., Leung, H.C.M., Yiu, S.M., Chin, F.Y.L., 2012. IDBA-UD: a de novo assembler for single-cell and metagenomic sequencing data with highly uneven depth. Bioinformatics 28, 1420-1428.
Pii, Y., Mimmo, T., Tomasi, N., Terzano, R., Cesco, S., Crecchio, C., 2015. Microbial interactions in the rhizosphere: beneficial influences of plant growth-promoting rhizobacteria on nutrient acquisition process. A review. Biology and Fertility of Soils 51, 403-415.
Prajapati, K., Sharma, M.C., Modi, H.A., 2013. Growth promoting effect of potassium solubilizing microorganisms on Abelmoscus esculantus. International Journal of Applied Agricultural Sciences 3, 181-188.
Rademacher, W., 2015. Plant Growth Regulators: Backgrounds and Uses in Plant Production. Journal of Plant Growth Regulation 34, 845-872.
Radzki, W., Gutierrez Mañero, F.J., Algar, E., Lucas García, J.A., García-Villaraco, A., Ramos Solano, B., 2013. Bacterial siderophores efficiently provide iron to iron-starved tomato plants in hydroponics culture. Antonie van Leeuwenhoek 104, 321-330.
Rehm, G., Schmitt, M.J.R.F., 2002. Potassium for crop production. Communication and Educational Technology Services, University of Minnesota Extension, pp. 2011.
Sayers, E.W., Agarwala, R., Bolton, E.E., Brister, J R., Canese, K., Clark, K., Connor, R., Fiorini, N., Funk, K., Hefferon, T., Holmes, J B., Kim, S., Kimchi, A., Kitts, P.A., Lathrop, S., Lu, Z., Madden, T.L., Marchler-Bauer, A., Phan, L., Schneider, V.A., Schoch, C.L., Pruitt, K.D., Ostell, J., 2018. Database resources of the National Center for Biotechnology Information. Nucleic Acids Research 47, D23-D28.
Senthilkumar, M., Swarnalakshmi, K., Govindasamy, V., Lee, Y.K., Annapurna, K., 2008. Biocontrol potential of soybean bacterial endophytes against charcoal rot fungus, Rhizoctonia bataticola. Current Microbiology 58, 288.
Shukla, P.J., Dave, B.P., 2018. Screening and molecular identification of potential exopoly-saccharides (epss) producing marine bacteria from the bhavnagar coast, gujarat. International journal of pharmaceutical sciences and research 9, 2973-2981.
Souza, R.d., Ambrosini, A., Passaglia, L.M.P., 2015. Plant growth-promoting bacteria as inoculants in agricultural soils. Genetics and Molecular Biology 38, 401-419.
Spence, C., Bais, H., 2015. Role of plant growth regulators as chemical signals in plant–microbe interactions: a double edged sword. Current Opinion in Plant Biology 27, 52-58.
Tu, Q., Lin, L., Cheng, L., Deng, Y., He, Z., 2018. NCycDB: a curated integrative database for fast and accurate metagenomic profiling of nitrogen cycling genes. Bioinformatics 35, 1040-1048.
Ullman, W.J., Kirchman, D.L., Welch, S.A., Vandevivere, P., 1996. Laboratory evidence for microbially mediated silicate mineral dissolution in nature. Chemical Geology 132, 11-17.
Verma, D., Palani, N., 1997. Manuring of tea in south India (revised recommendations). UPASI Tea Research Institute, India.
Vimal, S.R., Singh, J.S., Arora, N.K., Singh, S., 2017. Soil-plant-microbe interactions in stressed agriculture management: A review. Pedosphere 27, 177-192.
Vurukonda, S.S.K.P., Vardharajula, S., Shrivastava, M., SkZ, A., 2016. Enhancement of drought stress tolerance in crops by plant growth promoting rhizobacteria. Microbiological Research 184, 13-24.
Wahyudi, A.T., Astuti, R.P., Widyawati, A., Meryandini, A., Nawangsih, A.A., 2011. Characterization of Bacillus sp. strains isolated from rhizosphere of soybean plants for their use as potential plant growth for promoting rhizobacteria. Journal of Microbiology Indonesia 153.
Warnes, M.G.R., Bolker, B., Bonebakker, L., Gentleman, R., Huber, W.J.V.R.P.T.f.P.D., 2016. Package ‘gplots’.
Watanabe, F.S., Olsen, S.R., 1965. Test of an Ascorbic Acid Method for Determining Phosphorus in Water and NaHCO3 Extracts from Soil. Soil Science Society of America Journal 29, 677-678.
White, P.J., Karley, A.J., 2010. Potassium. In: Hell, R., Mendel, R.-R., Eds.), Cell Biology of Metals and Nutrients. Springer Berlin Heidelberg, Berlin, Heidelberg, pp. 199-224.
Yang, H., Wang, D., Li, B., Zeng, Z., Qu, L., Zhang, W., Chen, H., 2018. Effects of potassium salts loading on calcium oxide on the hydrogen production from pyrolysis-gasification of biomass. Bioresource Technology 249, 744-750.


